서 언
재료 및 방법
유전분석을 위한 집단 작성
TG10와 TG201의 Whole-Genome Resequencing 및 SNP 분석
Genotyping-by-sequencing을 이용한 유전지도 작성
호박 흰가루병 저항성 검정
흰가루병 저항성 QTL 분석
흰가루병 저항성 판정 분자표지 제작 및 검정
결과 및 고찰
호박 유전체 염기서열 및 변이 분석
호박 흰가루병 저항성 검정
애호박 유전 지도 작성 및 흰가루병 저항성 QTL 분석
가루병 저항성 분자표지 개발
흰가루병 저항성의 유전자 이입(introgression) 분석
서 언
박과는 118속 825종의 식물을 포함하는데, 호박속(Cucurbita genus)에는 15종이 알려져 있으며, 주로 C. maxima(서양계 호박), C. moschata(동양계 호박, 애호박), C. pepo(페포계 호박, 쥬키니) 3종이 식용으로 재배되고 있다(Lee et al., 2011). 국내에서는 주로 애호박과 쥬키니 호박이 생산되는데, 매년 9천 ha 이상의 면적에서 30만톤 이상이 생산되고 있다(KOSIS, 2020). 호박은 경제적으로는 물론 영양적으로도 중요한 작물이다(Weng and Sun, 2012).
국내 호박 흰가루병은 Podosphaera xanthii(syn. Podosphaera fusca, Sphaerotheca fuliginea)에 의하여 발생되는데, 박과 작물에 가장 만연하며 피해가 큰 절대 기생 곰팡이병으로 특히 쥬키니와 애호박에 그 피해가 크다(Chang et al., 2001). 흰가루병은 호박의 줄기, 엽병, 잎 표면에서의 하얀 균사체 증식 및 포자 형성으로 쉽게 알아볼 수 있는데, 감염 후 4 ‑ 7일 후에 나타난다. P. xanthii는 많은 종을 감염시키는데, 국화과, 박과, 꿀풀과, 현삼과, 가지과, 마편초과 등에서 중요한 경제작물을 포함하여 약 1만 종의 속씨식물에 병을 유발하며, 병으로 인한 볕 데임 증상, 불완전한 숙성, 저장성 부진 등을 야기하여 과실 수확 및 품질이 저하되고, 결국 백화 병변을 통한 광합성 저해로 식물체가 사멸된다(Glawe, 2008; PÉREZ-GARCÍA et al., 2009; Kim et al., 2014).
한편 최근에는 P. xanthii 자체에 대한 분자유전학적 분류 연구가 활발한데, 이 균은 대부분 대량의 분생포자를 통한 무성 번식으로 유전적 변이가 적다(Pirondi et al., 2015). 또한 기생근(haustoria) 전사체 분석에 의한 병원성 관련 유전자 탐색 및 genotyping-by-sequencing(GBS) 분석으로 균의 유전적 변이를 조사하였다(Polonio et al., 2019; Xiang et al., 2019).
호박 재배 중 흰가루병 방제는 일반적으로 살균제에 의존하는데(Chang et al., 2001; Moon et al., 2010), 오래 전부터 특히 살균제 투입이 많은 포장에서의 저항성 균의 출현이 살균제 사용에 장애가 되고 있다(O'Brien et al., 1988; O'brien, 1994; Lebeda et al., 2010; Vielba-Fernández et al., 2018). 따라서 살균제 이외에 자연 생산물과 오일(Shim et al., 2018), 길항기작을 갖는 세균과 진균(bio-agent) (Gafni et al., 2015; Hafez et al., 2018), 세균배양 부산물(Tanaka et al., 2017)을 사용하는 등의 다양한 생물적 방제가 시도되고 있다. 그러나 저항성 자원을 탐색하여 이용하는 유전·육종적 방법이 효과가 지속적이고 경제적이므로 가장 각광을 받고 있다(Cho et al., 2004; Cho et al., 2005; Kim et al., 2014; Holdsworth et al., 2016; Guo et al., 2018; Guo et al., 2019).
야생종 C. okeechobeensis subsp. martinezii와 C. lundelliana는 불완전한 단인자 우성 유전양상의 흰가루병 저항성을 갖고 있으며(Rhodes, 1964; Munger, 1976), 동양종 및 페포종과의 종간교잡이 가능한 것으로 오래 전부터 알려졌다(Contin, 1978; Cohen et al., 2003). 특히 국내에서는 동양종과 C. okeechobeensis subsp. martinezii와의 종간교잡을 통하여 흰가루병 저항성을 동양종 애호박으로 이입하여 품종 개발에 이용하였고(Cho et al., 2004; Cho et al., 2005), 개발된 흰가루병 저항성 애호박은 ‘천수’(출원-2001-57)와 ‘장수’로 품종 등록되었다. 국립원예특작과학원에서 품종 등록한 ‘광명’(출원-2004-11) 역시 같은 계통의 흰가루병 저항성 품종이다(Cho et al., 2009).
호박의 유전체 염기서열 분석은 C. pepo (Montero-Pau et al., 2018), C. maxima와 C. moschata(Sun et al., 2017)의 세 종에 대해 최근에 진행되었다. 유전체 정보는 유전체의 구조와 기능 및 진화 연구에 활용되고 있으며, 새로운 유전자의 탐색, 분자표지 개발과 육종에 응용 등에 도움이 되고 있다. 또한 유전체 정보를 기반으로 호박류에서 GBS를 응용한 연구가 활발한데(Chung et al., 2017), C. maxima의 F2 집단을 이용하여 GBS 방법으로 줄기 길이 특성을 매핑하였고(Zhang et al., 2015), C. pepo의 아종간 교잡의 RIL(recombinant inbred lines) 집단의 GBS 분석으로 조기 개화, 과일의 형태와 길이, 과피색과 과육색에 연관된 QTL을 탐색하였다(Montero-Pau et al., 2017). 또한 C. pepo의 pepo 아종과 ovifera 아종 7종의 whole-genome resequencing을 통한 비교분석으로 진화와 순화과정 중의 유전적 변이를 해석하고 형태적 변이와 관련된 유전자들을 추정하였다(Xanthopoulou et al., 2019).
본 연구의 목적은 종간 교잡으로 C. moschata에 이입된 흰가루병 저항성 유전자의 위치를 분석하고, 이의 유전자형을 예측할 수 있는 간편한 분자표지를 개발하여 새로운 품종 육성에 기여하는 것이다. 이를 위하여 우리는 유전체 염기서열, F2 집단 작성, GBS 분석, SNP 지도 작성 및 유전체 비교 등을 수행하고 그 결과를 제시하였다.
재료 및 방법
유전분석을 위한 집단 작성
홍익바이오(주) 보유 육종소재 중에서 순계로 정착된 흰가루병 감수성 계통 TG201(C. moschata)을 모계로 사용하고 저항성 계통 TG10을 부계로 사용하여 F2 집단을 작성하고 무작위로 선정된 204 개체를 흰가루병 저항성 유전분석에 사용하였다. TG10은 흰가루병에 저항성을 갖는 C. okeechobeensis subsp. martinezii 계통과 애호박(C. moschata)의 교잡 후대로 국내에서 개발된 계통에서(Cho et al., 2005) 유래된 육종소재이다.
TG10와 TG201의 Whole-Genome Resequencing 및 SNP 분석
애호박 육종소재 TG10(흰가루병 저항성)과 TG201(흰가루병 감수성)의 유전체 염기서열을 분석하였다. 염기서열 생산은 일루미나(Illumina) 체계를 이용하였다. 생산된 초기 데이터(raw data)에서 Trimmomatic을 이용하여 어댑터(adaptor)와 불량 염기서열을 제거하고, SOAPec을 이용하여 서열을 수정하였다. KmerGenie를 사용하여 유전체 조립을 위한 최상의 k-mer 값과 유전체 크기를 계산하고, SOAPdenovo2로 유전체를 조립한 후, Gapcloser를 사용하여 조립된 스캐폴드(scaffold)의 빈 곳 채우기를 반복적으로 수행하였다. 또한 조립과정에서 발생할 수 있는 부정확한 공통 염기서열 또는 삽입/결실(insertion/deletion, indel) 등을 교정하기 위하여 Pilon 프로그램으로 5회에 걸쳐 교정하였다. 교정이 완료된 TG10과 TG201 유전체 염기서열은 C. moschata 표준유전체를 대상으로 nucmer를 이용하여 정렬한 후 컨티그(contig)의 순서와 방향을 결정하고, 인위적으로 100개의 N을 삽입하여 컨티그를 연결하고 가상염색체(pseudomolecule)를 완성하였다.
단일염기다형성(SNP, single nucleotide polymorphism)과 삽입/삭제(indel)를 추출하기 위하여 애호박 T201의 유전체 염기서열을 기준으로 하고 BWA-mem를 이용하여 TG10의 염기서열을 정렬하고, 중복 염기서열의 영향 등을 보정한 후, GATK haplotypeCaller를 이용하여 SNP와 indel 변이를 추출하였다. Raw variant는 vcftools로 minimum depth 50, minimum genotype quality 20, no missing sample allowed의 조건으로 필터링하였고, TG201의 SNP 중에서 기준 유전자좌(reference allele)가 아닌 것을 제거하고, TG10에서 이형접합성(heterozygous) SNP를 제거하여 최종적으로 337,373개의 SNP을 선발하였다.
Genotyping-by-sequencing을 이용한 유전지도 작성
TG10과 TG201, 그리고 F2 204 개체의 잎에서 gDNA를 CTAB 방법으로 추출하고 제한효소 EcoR I과 Mse I을 사용하여 gDNA를 절단한 후 adapter를 붙여 라이브러리를 제작하였고, HiSeq 2000을 사용하여 염기서열 분석을 하였다. C. moschata 계통인 Rifu(Sun et al., 2017)를 참조 서열(reference sequence)로 BWA 0.7.12를 사용하여 alignment하였다. GATK UnifiedGenotyper를 통해 308,213개의 SNP을 추출하였고 TG10과 TG201에 다형성을 보이고 Qual 30, Depth 3 이상의 1,733개 SNP으로 필터링하였다. Sliding window approach로 5개의 근접한 SNP을 window로 설정하여 397개의 bin을 만들었다. Carthagene으로 logarithm of the odds(LOD) 5, distance threshold 30 cM의 조건으로 24개의 연관그룹을 만들었고 bin marker의 거리는 kosambi function으로 계산하였다.
호박 흰가루병 저항성 검정
상기의 유전분석 F2 집단과 대조 품종을 발아시킨 뒤, 직경 9cm, 토양 400mL의 포트에 정식한, 파종 후 10일 된 유식물체를 흰가루병 저항성 검정에 사용하였다. 식물체 재배는 온도 25 ± 5°C의 온실 조건에서 이루어졌다. 오이를 이용하여 계대배양한 흰가루병균(Podosphaera xanthii) 레이스 1을 접종원으로 사용하였다. 접종은 공기전염 방법을 사용하였는데, 접종할 호박 유식물체 포트 주변에 이병식물을 배치하여 공기전염이 되도록 하였다. 실험 중에 흰가루병 발생이 촉진되도록 수분 공급을 하였는데, 잎에 물이 닫지 않도록 주의하면서 포트에 직접 주었다. 저항성 판정은 파종 후 35일(접종 후 2주)에 제1 본엽과 제2 본엽에 발생한 흰가루병의 병반면적률(%) 및 포자형성 병반면적률(%)을 조사하였다. 저항성 판정기준은 병반면적률 또는 포자형성 병반면적률(%)에 따라서 0 ‑ 10%는 저항성, 11 ‑ 35%는 중도저항성, 36 ‑ 100%는 감수성으로 3단계로 평가하였다.
흰가루병 저항성 QTL 분석
Windows QTL Cartographer V2.5 프로그램을 이용하여 composite interval mapping을 실시하였다. 흰가루병 검정 자료는 병반발생률과 그 비율에 따른 저항성 유무(R/S) 및 포자형성률과 그 비율에 따른 저항성 유무(R/S)의 형식으로 모두 네 가지를 각각 분석에 사용하였다.
흰가루병 저항성 판정 분자표지 제작 및 검정
흰가루병 저항성 유전자좌가 위치한 TG10의 3번 염색체 86.2 cM(물리적 위치는 약 7.1Mb) 위치의 좌우 약 200 kb에 해당하는 영역에서 저항성인 계통 TG10, 감수성인 계통 TG201의 염기서열을 비교하여 저항성과 감수성을 구분할 수 있는 7개의 삽입/결실(indel, insertion/deletion) 분자표지를 제작하였다. 프라이머 디자인은 Primer3Plus(Untergasser et al., 2007)를 이용하였다. 각 분자표지의 흰가루병 저항성 판정의 유효성은 TG10과 TG201을 포함하여, F2 개체 및 시판 품종 등의 PCR 전기영동을 통하여 확인하였다.
결과 및 고찰
호박 유전체 염기서열 및 변이 분석
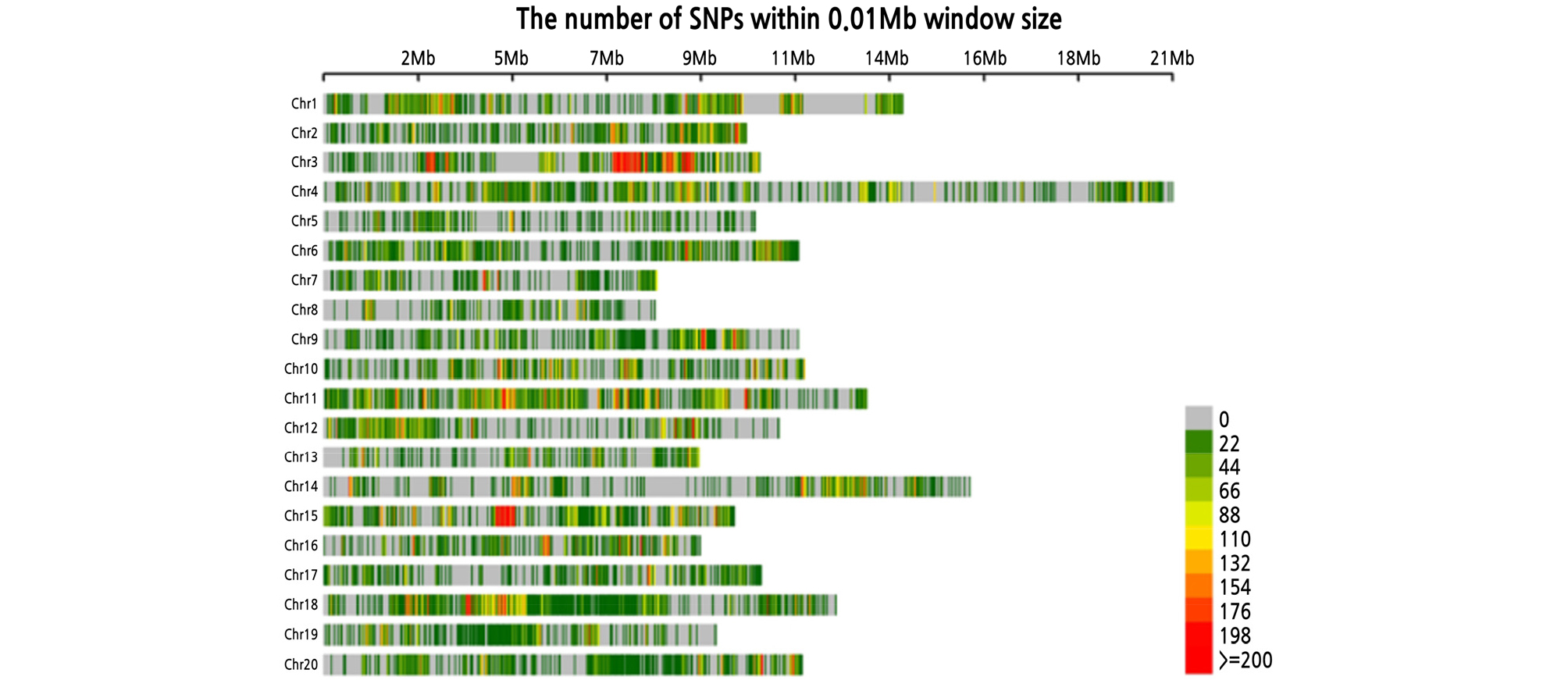
애호박 육종소재 TG201(흰가루병 감수성)과 TG10(흰가루병 저항성)의 유전체를 일루미나 방식으로 염기서열 분석하였다. TG201과 TG10의 조립된 유전체(NABIC 등록번호, NU-1206-000001 ‑ 000004와 NU-1207-000001 ‑ 000004) 길이의 합은 각각 232,213,264 bp 및 230,939,891 bp였으며, Mummer로 비교한 참조 유전체 ‘Rifu’ 크기의 97.3% 및 96.7%였다. TG10과 TG201에서 337,373개의 SNP을 추출하여 비교하였다. 선발된 TG201과 TG10간의 SNP의 분포는 염색체별로 차이가 컸는데, 3번 염색체가 40,127개로 가장 많았고(38 SNPs/10kb), 5번 염색체가 5,319개로 가장 적은 분포(5 SNPs/10kb)를 보여 7배 이상의 차이를 보였다. 염색체 3번의 7 ‑ 9Mb 지역과 15번의 4.5 ‑ 5.2Mb 지역의 경우 10kb 내에 200개 이상의 SNP가 밀집되어 있었다(Fig. 1). 염색체 5번, 7번, 8번, 13번, 17번, 19번과 같이 전체적으로 SNP 빈도가 특히 낮은 이유는 TG10과 TG201 모두 육종 과정 중에 특정 염색체 또는 재조합 블록(recombination block)을 공유하기 때문일 것으로 추정된다(Fig. 1).
호박 흰가루병 저항성 검정
TG201과 TG10의 F2 집단의 204개체의 유식물체에 흰가루병을 공기감염으로 접종하고, 2주 후에 병 발생 정도를 1엽과 2엽으로 구분하여 전체 병반면적률(%)과 포자를 형성하는 병반면적률(%)을 각각 측정하여 저항성 정도로 환산하였다. 저항성 정도는 3단계, 즉 저항성, 중도저항성, 감수성으로 구분하였고, 자료의 해석에서는 저항성과 중도저항성을 모두 저항성으로 간주하였다.
공시한 대부분의 식물체의 1엽에서 2엽에 비하여 상대적으로 병의 진전 정도가 심했는데, 병반면적률로 보면 1.38배이고 포자형성 병반면적률로 보면 1.56배 병 발달이 많았다. 따라서 저항성 판정 결과는 반대로 나타나는데, 2엽에 비하여 1엽에서 병반면적률에 의거해서는 약 76%, 포자형성 병반면적률에 의거해서는 약 89%로 나타났다.
공시 식물체 1엽과 2엽의 평균 병반면적률과 평균 포자형성 병반면적률에 의한 저항성:감수성 비율은 각각 57:43(1.3:1)과 78:22(3.5:1)로 나타났다(Tables 1 and 2). 즉 평균 포자형성 병반면적률에 의한 저항성 판정이 3:1(p > 0.05) 분리비를 보이는데, 이러한 검정 결과는 다른 연구에서도 C. moschata와 C. martinezii의 종간교잡 및 여교배 분석으로 흰가루병 저항성은 단인자 우성이며 일부 minor 유전자의 영향이 있다는 보고와 유사하다(Cho et al., 2005).
Table 1.
Evaluation of powdery mildew resistance by measuring the percentage of lesion area of leaf. Total 204 individuals of F2 between TG201 (susceptible) and TG10 (resistant) heirloom were assayed
Table 2.
Evaluation of powdery mildew resistance by measuring the percentage of spore bearing lesion area of leaf. Total 204 individuals of F2 between TG201 (susceptible) and TG10 (resistant) heirloom were assayed
호박 포장이나 하우스에서 흰가루병의 만연은 균사체 증식 후의 포자 형성과 산포가 중요한데, 흰가루병 저항성 품종의 경우 하엽에는 흰가루병이 발생해도 포자 형성이 심하지 않고 따라서 병의 전파가 잘 일어나지 않는 특성을 보인다. 호박의 흰가루병 저항성 계통은 진피세포벽 및 큐티클층의 두께와 상관이 있다는 보고가 있지만(Kim et al., 2014), 아직까지 흰가루병의 포자 형성 기작과 요인들이 연구된 결과는 없다. 그러나 병 검정 결과는 흰가루병 저항성은 포자 형성과 전파에 관련이 있으며, 저항성 판단은 포자 형성 병반면적률에 의거해야 함을 보여주고 있다.
애호박 유전 지도 작성 및 흰가루병 저항성 QTL 분석
흰가루병 검정에 사용된 TG10 x TG201 F2 집단 204 개체와 부모와 대조 계통을 포함하여 총 212 개체에 대해 GBS 분석을 하였는데, read 양이 <100 Mb보다 적은 12 개체와 SNP의 개수가 적은 6개체를 제외하고 194개체에서 1,733개의 SNP을 얻었다(Table 3). SNP 데이터를 사용하여 만들어진 bin은 393개이며 2,021cM 길이의 연관 지도를 작성하였다(Fig. 2). 2, 4, 6, 15번 염색체에서 연관 그룹이 2개로 만들어져서 C. moschata의 염색체 20개보다 많은 24개의 연관 그룹이 형성되었다. 특히 연관군 5번, 7번, 8번은 bin이 10개 이하였는데, 부모의 이 번호 염색체 염기서열 유사성이 높은 것이 원인이라고 판단된다(Table 4). 이 결과는 TG201과 TG10 유전체의 SNP 빈도에서도 같은 경향을 보였다(Fig. 1).
Table 3.
Summary of SNP calling and filtering from GBS data of 204 F2 individuals
| Filtering option | Number of SNPs | |
| Raw SNPsz | 308,213 | |
| SNPs corresponding to parents datay | 31,278 | |
| Final SNPsx | SNP quality ≥30 | 1,733 |
| Read depth of each SNP ≥3 | ||
| Genotype quality ≥30 | ||
Table 4.
Summary of SNP and bin information on each linkage group
흰가루병 저항성 QTL 분석은 병반면적률(%)과 포자형성 병반면적률(%)을 적용한 양적형질 분석과 각 면적률에 따른 저항성/감수성 판정에 따른 질적형질 분석의 4가지 방법으로 수행하였다(Tables 1 and 2). 전체적인 결과는 표현형 조사 형식에 무관하게 주요 QTL은 모두 연관군(염색체) 3번의 80 ‑ 102cM 범주에 들었으며, 주요 QTL의 최고치는 86.2cM에 위치했다. 이것의 물리적 위치는 TG10의 염색체 3번의 6.9 ‑ 7.3Mb 영역에 있었다. 이 QTL의 LOD 값은 12 이상이고 R2값은 16.3이다(Table 5 and Fig. 3). 한편 이러한 결과는 Holdsworth et al.(2016)이 C. okeechobeensis subsp. martinezii의 흰가루병 저항성 유전자좌 Pm-0를 C. okeechobeensis subsp. martinezii PI 532363 x C. moschata ‘Burpee's Butterbush’의 F2 연관군 10번에 매핑한 결과와 종간교잡에 의한 유전자 이입 방법과 이입된 유전체 단편의 내용이 유사한 것으로 판단된다.
Table 5.
Results of qualitative and quantitative analysis of powdery mildew resistant loci
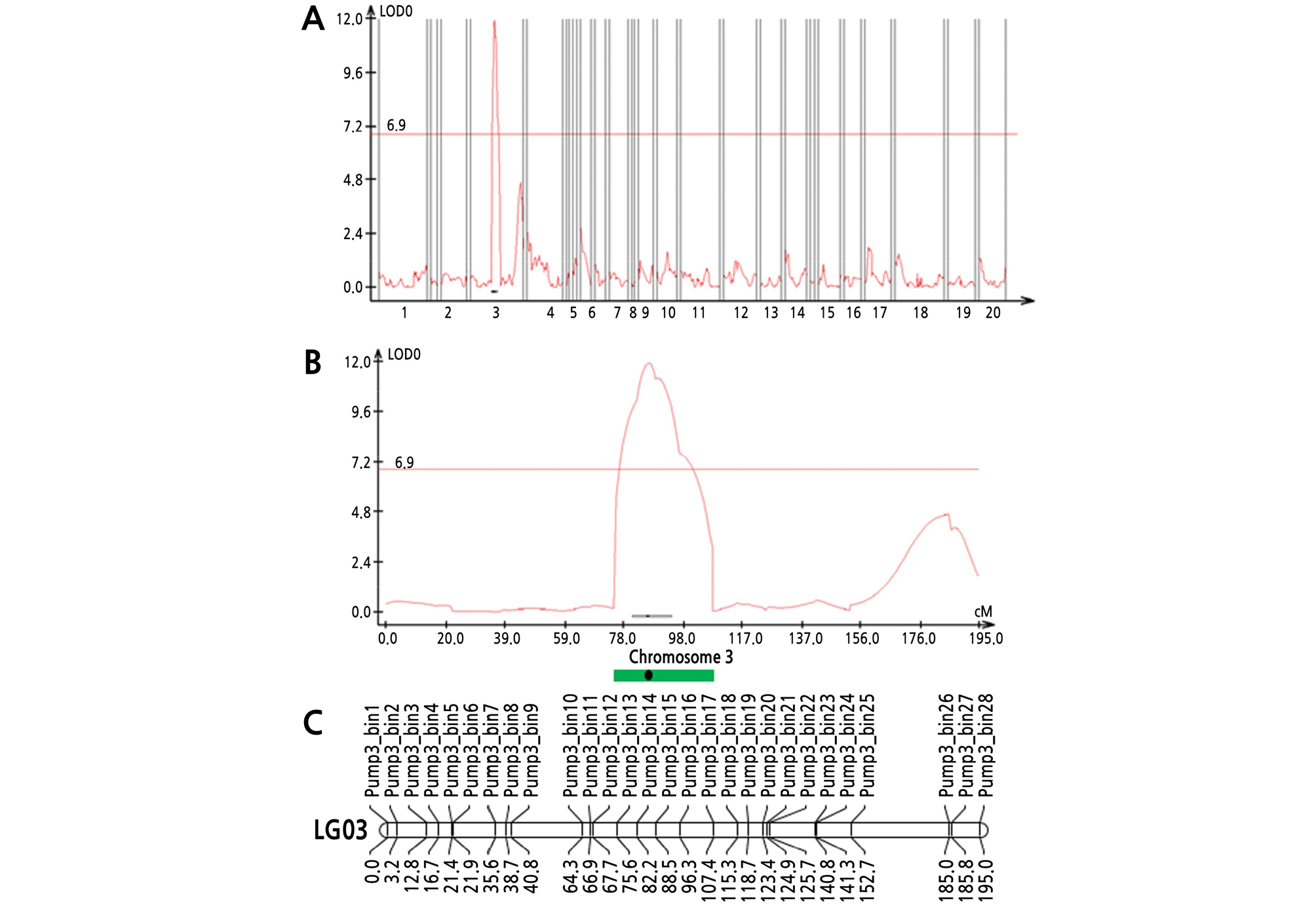
Fig. 3.
QTL mapping of powdery mildew resistance using the assay data of lesion area (%). (A) The major allele was located on linkage group 3. (B) An enlargement of data from A showing the peak position at 86.2 cM. (C) The major allele covers bins 14, 15, and 16 of linkage group 3. The green bar and black dot represent the region of the major allele and the peak position, respectively.
C. moschata에서 해당 QTL을 중심으로 500 kb 지역에 NBS-LRR 유전자를 포함하여 71 개의 유전자를 볼 수 있는데, 이 지역에서의 TG10과 TG201의 유전자 염기서열 및 유전자 발현 차이를 분석하는 등 일련의 분자생물학적 연구를 통하여 흰가루병 저항성 관련 유전자를 동정할 필요가 있다.
가루병 저항성 분자표지 개발
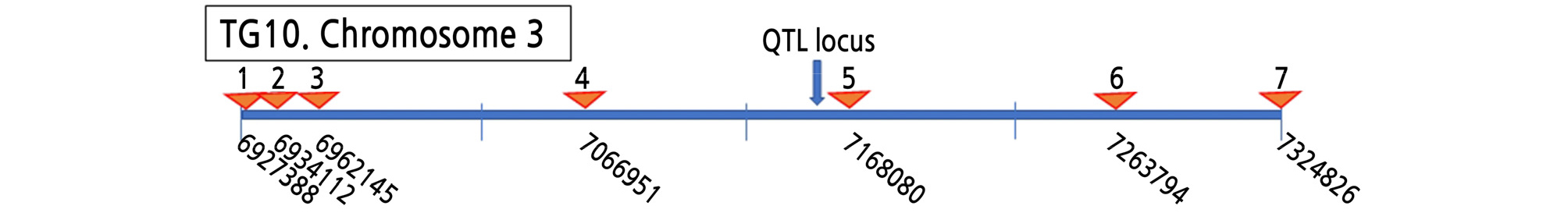
흰가루병 저항성이 위치한 3번 연관군에 해당하는 TG10(저항성) 염색체 3번의 6.9 ‑ 7.3Mb 부분의 염기서열과 TG201(감수성) 염색체 3번의 7.1 ‑ 7.5Mb 부분의 염기서열을 확보하고, ‘Rifu’의 해당 부분을 기준으로 염기서열을 비교하여, 저항성(TG10)과 감수성(TG201)을 구분할 수 있는 염기서열 삽입 및 결실(indel) 정보를 수집하였다. 수집된 indel 정보를 바탕으로 6,927,388bp에서 7,325,079bp까지의 약 400kb 영역에서 7개의 indel PCR 분자표지 세트를 제작하였다(Fig. 4 and Table 6).
Table 6.
Seven indel PCR marker sets for distinguishing resistant and susceptible trait in C. moschata varieties. Indel information was extracted by comparing the sequences around the mapping position of powdery mildew resistance on chromosome 3 of TG10 and TG201
제작된 흰가루병 저항성 PCR 분자표지(PMR)를 사용하여 TG10과 TG201의 DNA로 그 유효성을 확인하였다. 7개의 indel 분자표지의 PCR 산물은 각각 최소 21bp에서 182bp의 band 길이 차이로 아가로스 겔 전기영동상으로 저항성 계통과 감수성 계통의 다형성을 구분할 수 있었다(Fig. 5A). 이 외에 indel 분자표지들은 TG10 x TG201 F2 개체, 육종 소재 및 시판되는 호박 품종의 흰가루병 저항성을 판별할 수 있었다(Fig. 5B and 5C). Fig. 5C의 시판 품종 중 5종은 흰가루별 저항성밴드를 보이고, 품종 3은 흰가루병 감수성으로 동형접합성을 보인다.
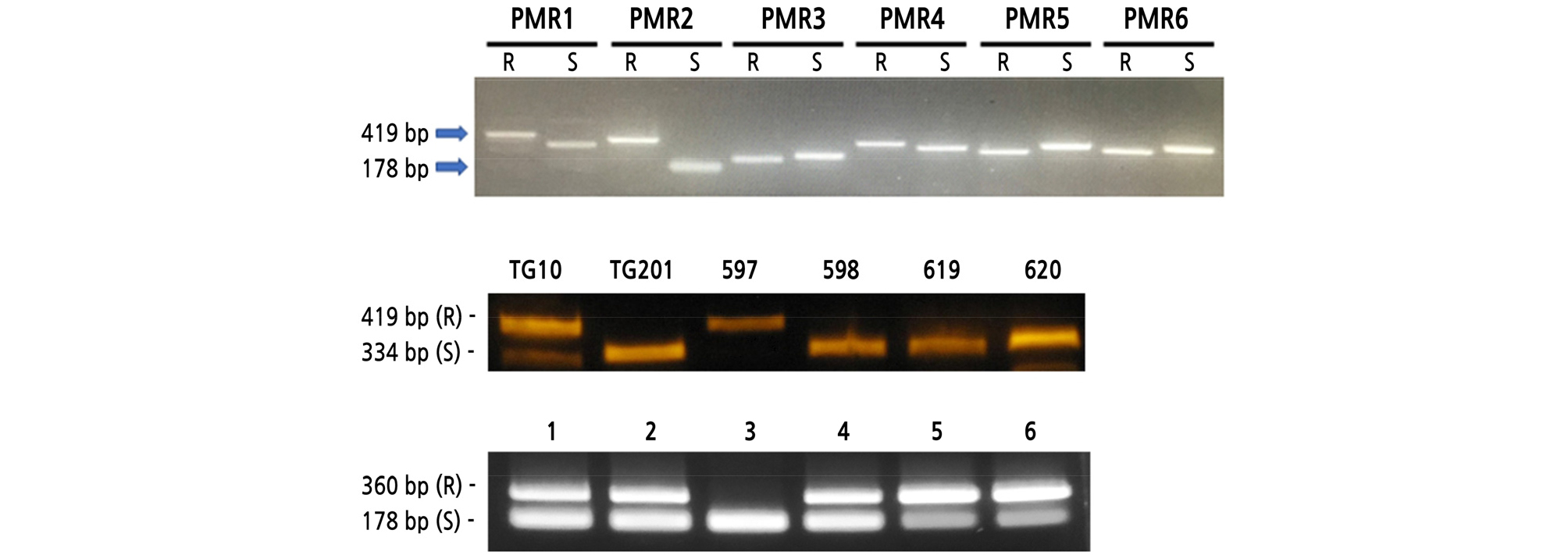
Fig. 5.
(A) PCR products of PMR markers (1-6) showing size difference between resistant (R=TG10) and susceptible (S=TG201) varieties. (B) Breeding materials were analyzed with primer PMR 1. (C) Commercial varieties were analyzed with primer PMR 2. Numbers 4 and 5 are the Hongikbio varieties. PCR products were separated on 1% agarose gel. Numbers on the left side show the size of PCR products. R, resistant; S, susceptible.
흰가루병 저항성의 유전자 이입(introgression) 분석
NCBI 데이터베이스에서 획득한 C. okeechobeensis subsp. martinezii의 유전체 염기서열 (accession no. SRX1292411)을 Trimmomatic 프로그램으로 Q score ≥20, min. length ≥36bp 조건으로 트리밍하여 약 403Mb 염기서열을 얻었다.
C. okeechobeensis subsp. martinezii로부터 TG10으로의 유전자 이입을 추정하기 위하여 C. okeechobeensis subsp. martinezii와 TG10은 동일하며 TG201과 다형성(polymorphism)을 보이는 즉, (martinezii=TG10)≠TG201 조건으로 GATK haplotypeCaller를 사용하여 총 89,165개의 SNP를 추출하였다. 이들 SNP는 3번과 15번 염색체에 그 수가 많았는데, 각각 18,511(전체의 20.7%)과 10,757(12.1%) 개였으며, 각각 염색체 3번의 6.8 ‑ 8.5Mb 영역과 염색체 15번의 4.2 ‑ 4.7Mb 영역에 뭉쳐서 나타났다(Fig. 6).
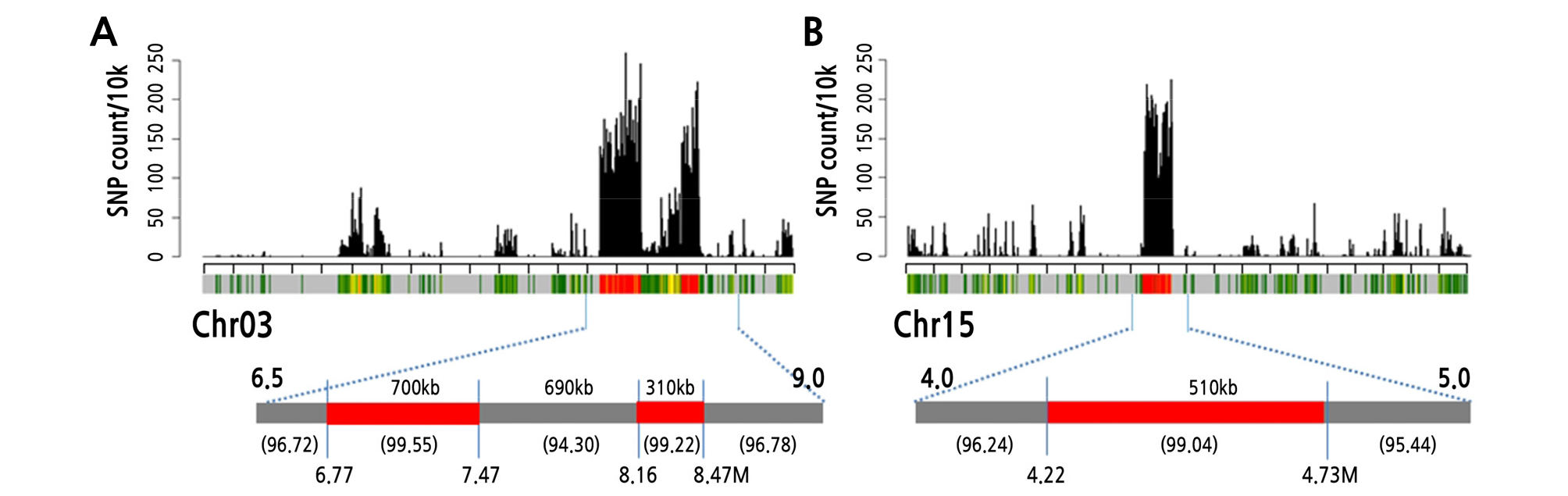
Fig. 6.
The (martinezii=TG10)≠TG201 SNP distribution across TG10 chromosomes 3 (A) and 15 (B). SNPs were counted and plotted in a 10-kb window. Red bars represent the proposed introgressions in chromosomes 3 and 15 of the TG10 genome. Numbers are the approximate block size in kilobasesand introgression position in megabases. Numbers inside parenthesare average % of sequence similarity between TG10 and C. okeechobeensis subsp. martinezii.
염색체 3번의 (martinezii=TG10)≠TG201한 부분(붉은 색 부분)의 뚜렷이 높은 SNP 빈도는 이 지역의 염기서열을 TG10과 martinezii가 공통적으로 갖고 있으며, TG201 및 ‘Rifu’의 염기서열과는 많이 다르다고 볼 수 있다(Fig. 6A). 다른 한편으로는 TG10과 martinezii 사이의 염기서열 유사성은 (martinezii=TG10)≠TG201 지역(붉은 색 부분)에서 99% 이상을 보이는 반면, 그 외의 지역에서는 94 ‑ 96%로 큰 차이를 보인다. 이상의 결과는 염색체 3번의 6.77 ‑ 7.47Mb와 8.16 ‑ 8.47Mb, 만 TG10과 martinezii가 공유하고 있으며, 즉 이 부분이 유전자 이입된 부분이라 수 있다(Fig. 6B). 또한 이와 같은 염기서열 양상을 보이는 염색체 15번의 4.22 ‑ 4.73Mb 영역도 martinezii로부터 이입된 부분이라 할 수 있었다.
C. moschata F2 집단을 통해 매핑한 흰가루병 저항성 유전자의 위치와 TG10의 염색체 3번에 이입된 C. okeechobeensis subsp. martinezii의 지역이 중복되므로 TG10의 염색체 3번에 이입된 유전 지역은 Pm-0을 포함할 것으로 추정한다. Holdsworth et al.(2016)이 C. pepo의 유전체를 참고하여 작성한 Pm-0에 연관되어 있는 두 CAPS 분자표지와 마찬가지로 7개의 indel 분자표지 또한 흰가루병 저항성을 구분 지을 수 있다. Indel 분자표지는 C. moschata ‘Rifu’와 TG10의 유전체를 참고 서열로 작성되었기에 이를 바탕으로 Pm-0의 위치를 좁혀 나갈 수 있을 뿐만 아니라 흰가루병 저항성 애호박 소재를 육성하는 데 활용할 수 있을 것이다.