서 언
재료 및 방법
식물 재료
샘플링 및 DNA 추출
ms1035 유전자 연관 마커를 이용한 foreground 선발
Background 선발을 위한 SNP 마커 선별 및 유전자형 분석
표현형 선발
통계분석
결 과
웅성불임 엘리트 계통육성을 위한 MR10-3211 계통의 특성
Foreground 선발을 위한 AAD 마커의 유효성 분석
BC1F1 세대의 유전자형 분석
BC2F1 세대의 유전자형 분석
BC2F2 및 BC2F3세대에서 유전자형 분석
고 찰
서 언
과학기술의 발달과 더불어 식물 육종기술은 커다란 진보를 보였으며, 특히 포스트 게놈시대를 맞아 다양한 기술을 이용하여 품종 육성을 하고 있다. 작물의 품종 개발 과정에서는 교배 등을 통해 웅성불임, 수량, 과색, 과형 및 스트레스 저항성 등과 같은 형질 관련 유전자의 엘리트 계통을 육성하고 있다(Vidal et al., 2012; Jang et al., 2015). 이와 같은 형질변이의 대부분은 유전변이로서 교배 등 다양한 육종방법을 통해 특정 계통에 전달시켜 분리되지 않도록 고정하여 이용하고 있다. 특히 웅성불임은 기능적으로 약, 화분 또는 웅성배우자를 생산하지 못하는 현상을 말한다. 식물의 웅성생식 기관 형성은 먼저 포자형성세포에서 웅성원기가 만들어져서 융단세포(tapetum) 및 소포자 모세포(microspore mother cells)를 형성한다. 그 후, 감수분열을 통해 1배체 소포자를 형성하고 융단조직에 의해 분비되는 callase의 활성에 의해 소포자로 분리되어 유사분열을 통해 성숙화분이 방출하는 단계로 구성되어 있다(Goldberg et al., 1993; Zhang and Yang, 2014). 웅성불임성은 웅성생식기관 형성과정에서 이상이 생기게 되면 기능적으로 웅성배우자 역할을 할 수 없어 종자를 생산할 수 없다(Chase et al., 2010; Chen et al., 2019). 진화적인 측면에서 보면 식물의 웅성불임성은 자가수정을 방지하고, 타가수정을 통해 종의 유전적 다양성을 높이는 수단이다(Hanson, 1991).
토마토(Solanum lycopersicum L.)는 가지과(Solanaceae)에 속하는 대표적인 채소작물로서, 국내뿐만 아니라 전세계적으로 생산량 및 소비량이 많아 시장에서 높은 경제적 가치를 가진다. 특히 토마토는 Agrobacterium을 통한 형질전환이 가능하고, 비교적 짧은 생활사 및 작은 유전체 크기를 가지고 있어 가지과 작물의 육종 모델로 사용되어 왔다(Foolad, 2007). 최근 토마토의 유전체 해독이 완료되고(The Tomato Genome Consortium, 2012), NGS(Next generation sequencing)를 이용한 대용량 염기서열 분석이 가속화되면서 육종에서 유전체 기반의 분자 육종이 갖는 중요도는 더욱 더 커지고 있다. 토마토 종자는 대부분 F1 품종이 상업적으로 이용되고 있으며, 인공수분에 의해 채종되기 때문에 종자생산 가격이 높고, 자가수정된 종자 혼입에 따른 F1 순도에 문제가 되고 있다(Bhatt et al., 2001; Atanassova, 2007; Solieman et al., 2013).
이런 불리한 점을 극복하기 위하여, ps2, ms10 및 ms32와 같은 토마토 웅성불임 유전자를 이용하여 F1 종자 생산에 이용하려는 노력을 많이 하고 있다(Cheema and Dhaliwal, 2005; Gardner and Panthee, 2010; Panthee and Gardner, 2013). 이중 ps2 유전자는 polygalacturonase를 암호화하고 있는 PS2 유전자의 구조적 변이에 의해 얻은 것이며, ms10 유전자는 bHLH 전사인자(transcription factor) 관련 유전자 프로모터 부분에 398bp 단편이 삽입되어 유전자 발현을 억제시킴으로써 웅성불임성을 나타낸 것으로 알려졌다(Gorguet et al., 2009; Jeong et al., 2014). 또한 웅성불임성 관련 ms10 유전자는 배축색에 관여하는 안토시아닌 합성 관련 aa 유전자와 매우 밀접하게 연관되어 있다고 보고하였다(Mutschler et al., 1987). 따라서 웅성불임성 개체는 어린 육묘의 배축에 안토시아닌 결손(anthocyanin absent, aa)과 같은 형태적 마커의 도움으로 쉽게 동정 가능하였다(Cheema and Dhaliwal, 2005; Atanassova, 2007; Gardner and Panthee, 2010; Panthee and Gardner, 2013).
그러나 aa는 열성유전자에 의해 지배되는 것으로 이형접합형 상태가 되면 표현형으로 구분할 수 없기 때문에 여교배 후, 자식이나 검정교배 과정을 더 거쳐야 한다. aa 유전자의 분자표지는 여교배 후대에서 동형접합형 및 이형접합형을 구분할 수 있어 육종연한을 획기적으로 단축시킬 수 있다고 하였다(Zhang et al., 2016). 분자표지를 여교잡 육종에 적용하는 maker- assisted backcrossing(MABC)은 분자표지를 이용하여 유묘기에 여교잡 자손의 염색체 조성을 확인함으로써 신속하고 정확하게 반복친의 염색체 이입개체를 선발할 수 있어 육종 연한과 비용, 노력 등을 효율적으로 감소시킬 수 있다고 하였다(Servin and Hospital, 2002; Collard and Mackill, 2008). 따라서 효율적으로 MABC법의 적용을 위해서는 foreground 마커, background 마커의 염색체상 위치 및 수, 여교배 횟수에 의해 좌우된다고 하였다(Herzog and Frisch, 2011).
본 연구에서는 상업적으로 이용하고 있는 엘리트 MR10-3211 계통에 ms1035 유전자를 안정적으로 도입하고자, foreground 마커는 Mutschler et al.(1987)이 보고한 aa 마커를 사용하였고, background 마커는 Jung et al.(2016)이 보고한 resequencing data 유래 SNP 마커를 선발하여 유효성 실험을 거쳐 이용하였다. 따라서 MABC법을 이용한 토마토 웅성불임 여교배 육종 프로그램은 엘리트 MR10-3211 계통의 게놈 조성에 ms1035 대립유전자의 이입개체를 신속하게 선발함에 따라 육종연한 및 소요비용을 획기적으로 감소시켰다.
재료 및 방법
식물 재료
본 연구에 사용한 토마토 엘리트 계통 MR10-3321은 (주)토마토연구소에서 상업용 F1 품종육성에 모친계로 이용하고 있는 복합내병성 계통이다. 웅성불임 계통 T2-517은 농촌진흥청 국립원예특작과학원에서 ms1035 유전자를 가지고 있는 것을 분양받았다. F1 식물은 T2-517(donor parent) 계통을 모친으로, MR10-3321(recurrent parent)을 화분친으로 교배하였다. 그리고 BC1F1 종자는 F1 식물을 MR10-3321와 여교배하였다. BC1F1 세대에서 ms1035 유전자를 위한 foreground 선발은 Zhang et al.(2016)이 보고한 배축에 안토시아닌 결손 관련 AAD 분자표지를 이용하였다. BC1F1 세대에서 목표 유전자(Aa)를 가지며, 반복친 게놈이 가장 높게 background 마커가 회복되고, 표현형적으로 가장 유사한 식물체를 선발하여 MR10-3321으로 여교배하여 BC2F1 종자를 육성하였다. 각각의 여교배 세대에서는 엘리트 계통과 함께 foreground, background 및 표현형 선발을 수행하였다. 또한 BC2F1에서 선발한 개체는 BC2F2 및 BC2F3 세대를 진전시켜 최종 육종소재로 이용하였다. 모든 토마토 육묘는 50공 트레이에 멸균한 토양을 이용하여 27°C에서 18시간 명조건(265µmol·m-2·s-1) 및 18°C에서 6시간 암조건의 생장상에서 생육시킨 후, 8매 잎 상태일 때 한경대학교 원예생명과학과 온실에 옮겨 생장시켰다.
샘플링 및 DNA 추출
DNA 추출을 위한 잎 샘플링은 발아 후 21일 육묘로부터 수행하였다. 게놈 DNA는 CTAB(cetyltrimethyl ammonium bromide)법을 사용하여 추출하였다(Saghai-Maroof et al., 1984). DNA 농도는 Nanodrop spectrophotometer(NanoDrop Technologies, Wilmington, DE, USA)로 측정하였으며, 최종 농도는 TE buffer(pH 7.0)을 이용하여 20ng·µL-1로 조정하여 사용하였다. DNA의 순도 및 양의 검토는 1% agarose 겔에서 전기영동을 통해 확인하였다.
ms1035 유전자 연관 마커를 이용한 foreground 선발
Zhang et al.(2016)은 ms1035 유전자와 매우 밀접하게 연관되어 있는 AAD 분자표지를 보고하였다. AAD 마커는 3개 프라이머(AAD-F, 5’-GGGACAAAATACAACGACGAC-3’; AAD-R1, 5’-GCTGATGTTGATGTGAAGGAA-3’ 및 AAD-R2, 5’-TAAAAGGGCTAGTGACTTGGTGT-3’)로 구성되어 있다. PCR 조건은 94°C에서 4분간 1 cycle, 94°C에서 30초, 55°C에서 30초, 72°C에서 40초 조건으로 32 cycles 수행한 후, 마지막으로 72°C에서 5분간 1 cycle 수행하였다. PCR 산물은 1.5% agarose 겔에서 전기영동하여 확인하였다.
Background 선발을 위한 SNP 마커 선별 및 유전자형 분석
Background 선발을 위해 사용한 SNP 마커세트는 Jung et al.(2016)에 의해 보고한 국내 토마토 엘리트 계통들을 기반으로 10X 이상의 NGS 리드를 생산하여 계통간 SNP 변이로부터 선발하였다. 선발 방법은 맵핑된 리드들을 SNP 콜링(calling)의 정확도를 높이기 위해, 게놈 분석 툴인 GATK(http://www.broadinstitute.org/gsa/wiki/index.php/The_Genome_Analysis_ Toolkit)를 이용하여 이중 마스킹(duplicate masking), 국소적 재정렬(local realignment) 과정을 수행한 후 GATK-Unified Genotyper(DePrisrto MA et al., 2011)를 이용해 SNP와 InDel을 예측하였다. 계통들에서 만들어진 SNP/InDel을 염색체의 좌(loci)를 기반으로 병합하고 SNP/InDel 매트릭스(matrix)를 생성한 후, 각 SNP 위치좌(locus)를 대상으로 인접 60bp에서 SNP 또는 InDel이 나타나는지 유무를 조사하였다. 인접 60bp에서 SNP나 InDel이 없는 부분을 SNP 마커 개발의 타겟으로 선정하고 Primer3(http://bioinfo.ut.ee/primer3-0.4.0/primer3/input.htm)를 이용하여 특유의 SNP를 검출할 수 있는 프라이머를 이용하여 단일 위치 좌만 증폭하는지를 평가한 후 단일 염기좌 맵핑 영역만 이용하여 HVR(hyper-variable region)을 발굴하였다. 이로부터 11Mb-23Mb 단위로 48개의 SNP 마커를 임으로 선정하여 Fluidigm® EP1TM system(Fluidigm, USA)에 사용하였다. 각 양친과 여교배(BC1F1, BC2F1 및 BC2F2) 세대의 유전자형 분석은 EP1TM의 48 × 48 Dynamic Array Chip을 이용하여 유전자형을 분석하였다(Suppl. Table 1s). 모든 분석방법은 Fluidigm사에서 제공한 실험 매뉴얼에 따라 수행하였으며, Fluidigm 플랫폼은 순천대학교 골든씨드 프로젝트 원예종자사업단에서 실시하였다.
표현형 선발
목표유전자(Aa)가 이입되고 반복친 MR10-3211에 가장 유사한 표현형을 선발하기 위해 토마토 표준재배법을 이용하여 온실에서 과일이 성숙할 때까지 재배하였다. 표현형 선발은 foreground 선발 후, BC1F1, BC2F1 및 BC2F2 세대의 모든 개체별로 평가하였다. 표현형의 특성은 초형, 숙기, 과형, 과중, 과색, 어깨색, 경도, 내병성 등의 특성을 조사하였다. 선발한 식물체들은 MR10-3211 계통과 유사 정도에 따라 계급화하였으며, 여교배 종자는 표현형 계급이 가장 높은 개체를 이용하여 수확하였다.
통계분석
Foreground 선발에서 AAD 마커로 분석하여 얻은 결과는 MR10-3211 유래 AA형, T2-517 유래 aa형, 이형접합 상태 Aa형으로 표시하였다. 카이자승 분석은 공식, Chi2 = Σ(O−E)2/E, 여기에서 O는 관찰치, E는 기대치 값으로 계산하였다. Background 선발에서 마커로부터 얻은 데이터는 Graphical Genotyper(GGT 2.0) 소프트웨어에 의해 분석하였다. 반복친과 같은 동형접합형, 공여친과 같은 동형접합형, 그리고 이형접합형은 각각 A, B 및 H로 표시하였다. 이들 데이터는 Microsoft Excel file에 넣어 GGT 2.0 소프트웨어에 의해 분석하여 반복친 동형 대립유전자형 퍼센트(% A), donor 동형 대립유전자형 퍼센트(% B), 이형 대립유전자형 퍼센트(% H)를 계산하였다. 각 세대의 식물체로부터 표현형은 조사항목에 따라 측정하여 평균값으로 표시하였다.
결 과
웅성불임 엘리트 계통육성을 위한 MR10-3211 계통의 특성
본 연구에서는 웅성불임 계통육성을 위해 MR10-3211 엘리트 계통(반복친)의 생리·생태적 특성은 Table 1과 같다. MR10-3211 엘리트 계통은 무한생장형으로 제1화방의 꽃이 피는 시기가 비교적 빠른 조생종이며, 화방발달이 1단, 2단까지는 단화방이 많으며 3화방 이후부터 다화방으로 발달한다. 꽃은 노란색이며 꽃자루는 손으로 수확이 용이한 이층이며, 평균과중이 230.2g으로 고구형이다. 성숙 전 어깨색은 옅은 녹색이며 착색은 핑크색이고 당도는 6.0°Brix 이다. 또한 MR10-3211 계통의 내병성은 토마토황화잎말림 바이러스(TYLCV)의 Ty1, Ty3 유전자, 토마토모자이크 바이러스(ToMV)의 Tm2a 유전자, 시들음병(Fusarium)의 F2 유전자, 근부위조병(Fusarium radicis)의 Fr 유전자, 뿌리혹선충(Nematode)의 N 유전자, 반신위조병(Verticillium)의 Ve 유전자, 잎곰팡이병(Cladosporium)의 Cf 유전자, 토마토 잎마름 역병(phytophthora infestans)의 Ph3 유전자 및 토마토 반점시듦병(spotted wilt virus)의 Sw5 유전자 등을 지니고 있는 복합내병성 계통이다(Table 1).
Table 1. Morphological characteristics and disease resistance of MR10-3211 and T2-517 tomato inbred lines used for marker-assisted backcrossing
yFirmness: MF (medium firm), F (firm), FF (very firm).
xDisease resistanc: Ty1 and Ty3 (Tomato yellow leaf curl virus), Tm2a (Tomato mosaic virus), F2 (Fusarium race 2), F3 (Fusarium race 3), Fr (Fusarium radicis), Ve (Verticilium), N (Nematode), Cf (Cadosporium), Ph3 (Phytophthora infestans), Sw5 (Tomato spotted wilt virus).
Foreground 선발을 위한 AAD 마커의 유효성 분석
F1 종자는 MR10-3211 계통과 T2-157 계통간 교배를 통해 생산하였으며, F1 식물체는 자가수정에 의해 F2 종자를 육성하여 이용하였다. F1 및 F2 세대에서 AAD 마커의 foreground 선발을 위한 유효성 분석을 조사한 결과 Fig. 1과 같다. AAD 마커는 T2-517 계통의 녹색의 배축색(aa), MR10-3211 계통의 자색의 배축색(AA) 및 F1 잡종 식물체의 자색의 배축색(Aa)의 유전자형을 잘 구분할 수 있었으며, F2 집단에서 1: 2: 1로 분리되었다. 또한 분리된 식물체의 불임성 조사를 한 결과 녹색의 배축을 가진 aa 식물체들에서 모두 불임으로 나타났다. 따라서 AAD 마커가 웅성불임식물체 육성을 위해 여교배 후, foreground 선발 마커로 활용 가능하다고 판단되었다.
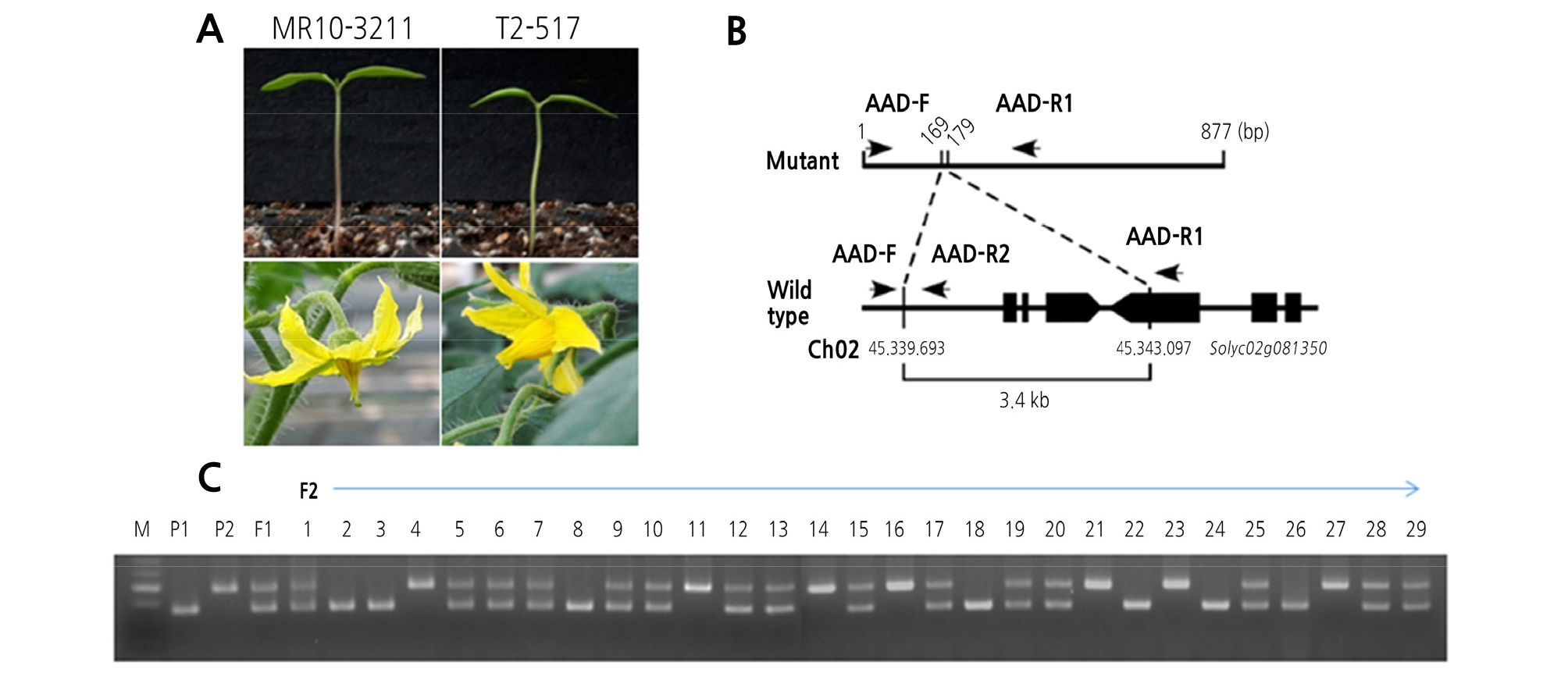
Fig. 1.
Validation analysis of AAD markers in foreground selection for the MABC program. A: Images of seedlings and flowers of wild-type tomato line MR10-3211 (left) and the aa mutant T2-517 (right), B: The aa locus harbored a 3.4-kb deletion. This deletion resulted in the loss of the entire gene and part of the Solyc02g081350 gene. AAD-F, AAD-R1, and AAD-R2 were the primers used in generating the AAD marker, C: Agarose gel electrophoresis of PCR fragments amplified from MR10-3211, T2-517, F1, and F2 plants using the AAD marker.
BC1F1 세대의 유전자형 분석
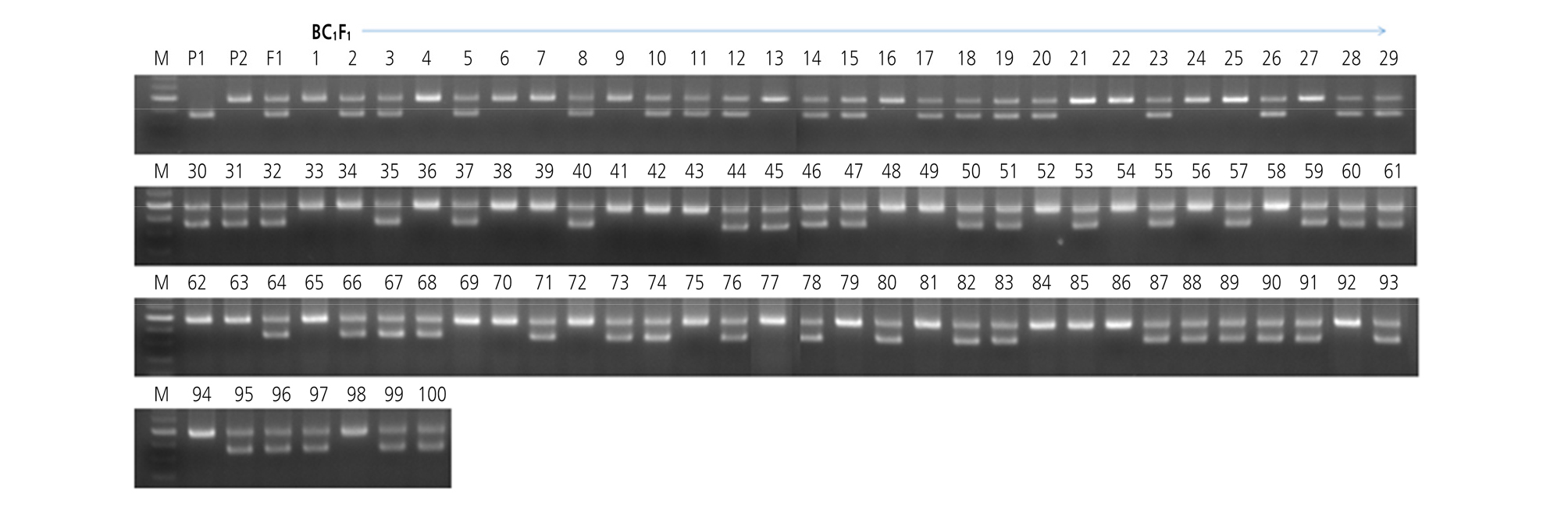
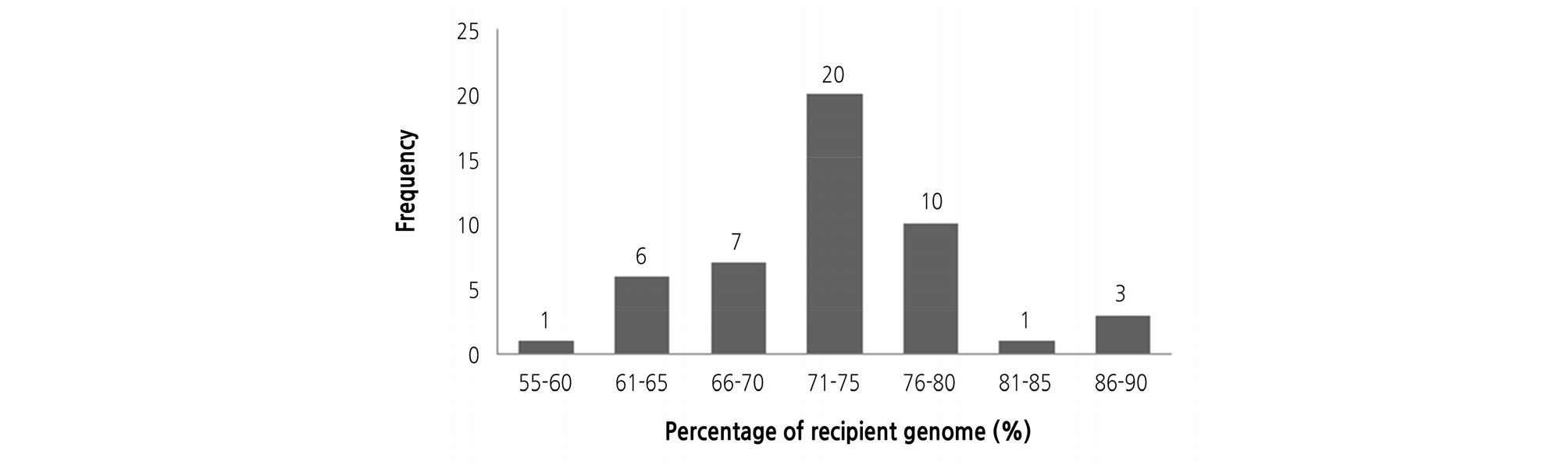
Foreground 선발을 위해 이형접합체를 가진 F1 식물은 MR10-3211 계통과 여교배하여 100개의 BC1F1 식물체를 육성하였다. BC1F1 세대에서 AAD 마커를 이용하여 foreground 선발한 결과는 Table 2와 같으며, 카이자승 실험을 통해 BC1F1 세대에서 1:1으로 잘 분리되었다(Table 2). 조사한 100개의 BC1F1 식물체 중에서 ms1035 유전자에 밀접하게 연관된 AAD 마커 유래 Aa 형은 58개로 확인되었다(Fig. 2). 선발한 58개 식물체들은 포장에 옮겨 표현형을 조사한 결과 웅성가임으로 Msms 유전자형이라고 간주하고 다음 실험을 진행하였다. Background 선발을 위한 SNP 마커 세트는 Jung et al.(2016)에 의해 보고한 resequencing 결과를 토대로 MR10-3211 계통과 T2-517 계통 간 다형을 보이는 SNP 마커를 염색체 별 4개씩 총 48개를 선정하여 염색체 상에 mapping하였다(Table 3, Suppl. Fig. 1s). Foreground 선발을 통해 얻은 58개 중에서 48개의 BC1F1 식물체를 대상으로 background 분석을 실시한 결과, 반복친 게놈(RPG) 회복 비율은 59.6 - 87.2%의 범위였으며, 평균 72.8%를 보였다(Fig. 3). AAD 마커에 의해 선발한 T2-517 공여친 유래 단편은 염색체 2번에 속해 있었으며, 염색체 1, 8, 11번은 다른 염색체와 비교해 볼 때 선발된 식물체들에서 거의 대부분이 회복되었다(Fig. 4). BC1F1 세대에서 목표유전자 Aa 유전자형을 가지며, 48개의 SNP 세트에서 MR10-3211 계통의 유전자형(RPG) 회복률이 가장 높은 식물체는 N3, N4 및 N20으로 87.2%를 보였다(Fig. 3). 따라서 foreground, background 선발 및 표현형에 입각하여 recurrent parent genome, RPG 회복률이 높으면서 MR10-3211 계통의 표현형과 유사한 BC1F1 식물체를 선발하여 BC2F1 세대를 육성하였다.
Table 2. Genotyping results of the AAD marker in F2, BC1F1, BC2F1, and BC2F2 generations derived from MR10-3321 (recurrent) x T2-517 (donor)
Table 3. Information of SNP markers used for background selection
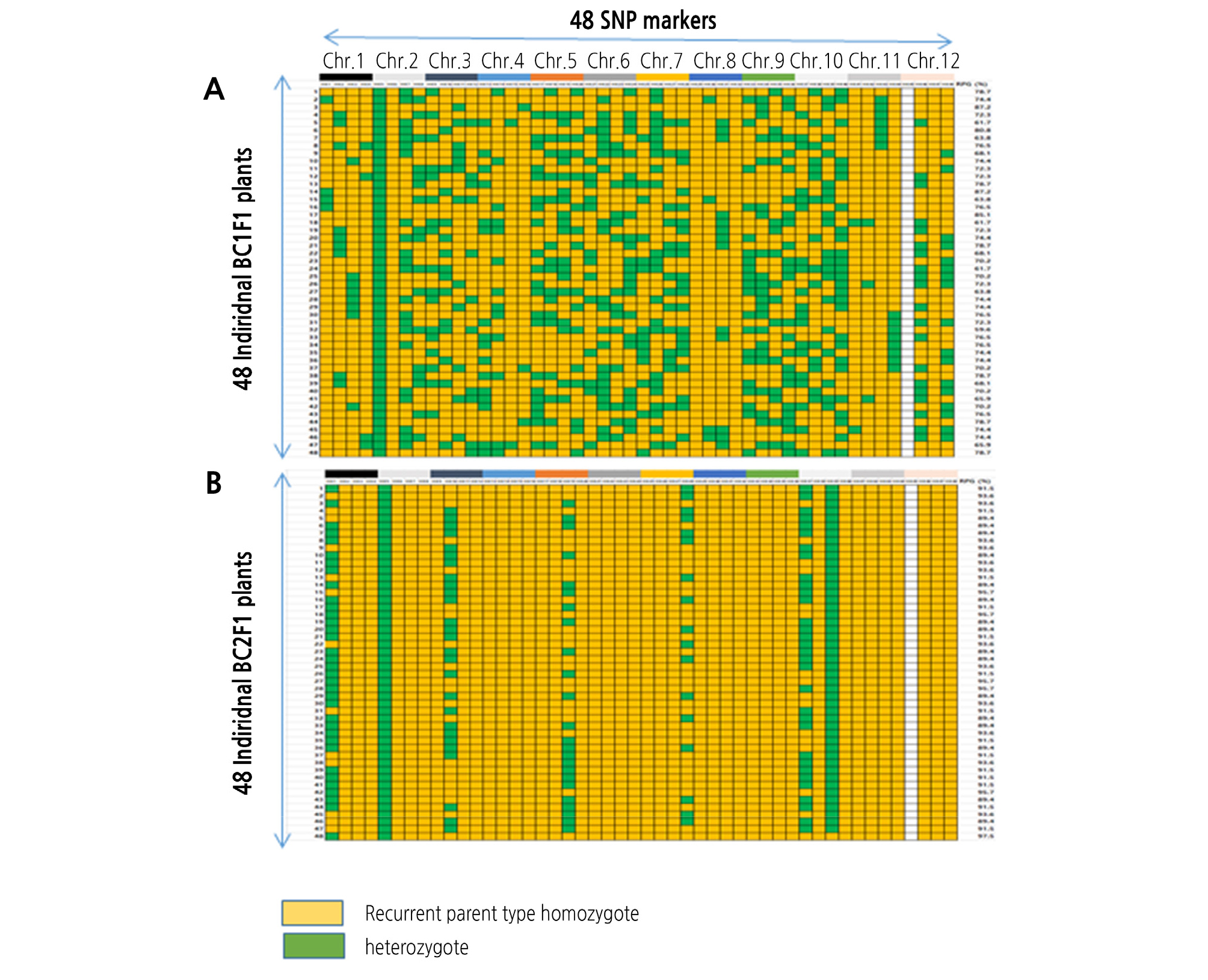
Fig. 4.
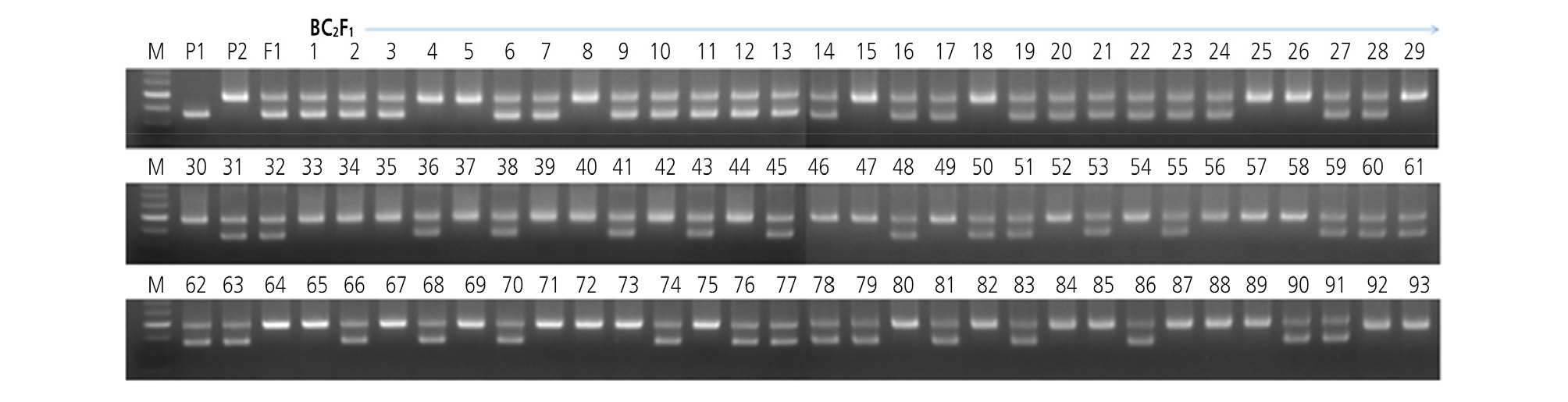
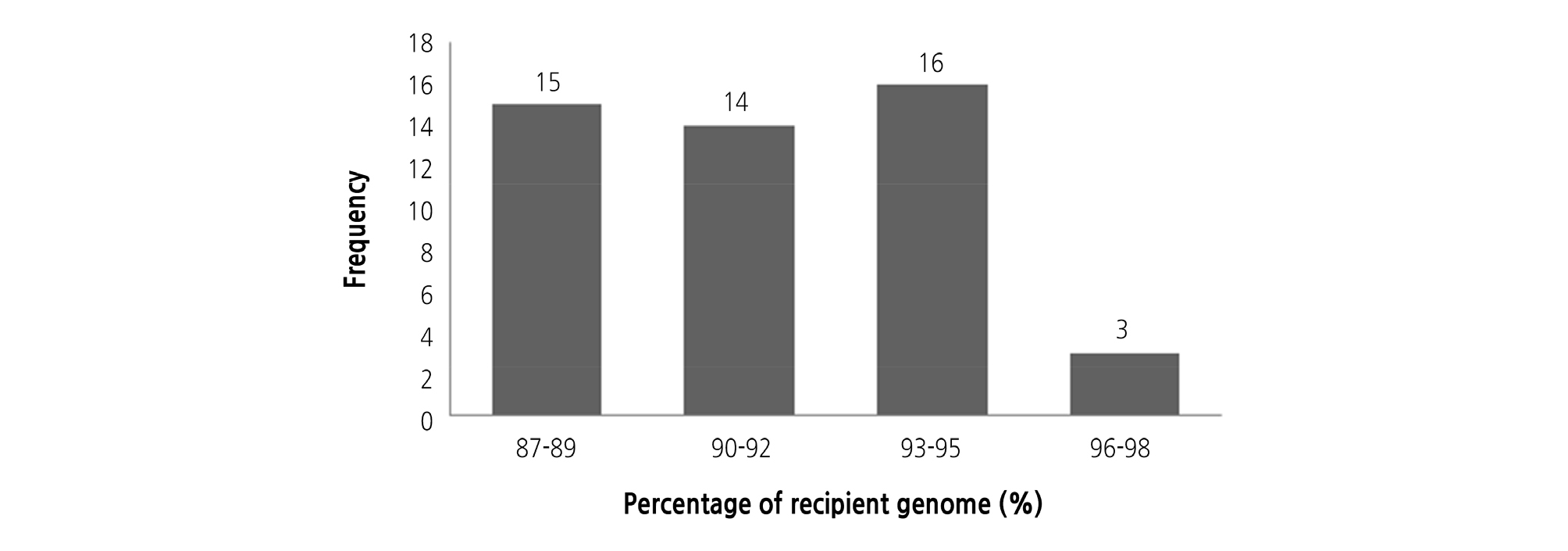
Genotyping results of the background selection in the backcross populations of the tomato. A: Genotyping results of the BC1F1 population. A total of 48 individuals and 48 SNP markers were used. The highest recovery rate was 87.2% and the lowest recovery rate was 59.6%; B: Genotyping results of the BC2F1 population. A total of 48 individuals and 48 SNP markers were used. SNP markers homozygous in BC1F1 were not duplicated with markers used in BC2F1. The highest recovery rate was 97.8% and the lowest recovery rate was 89.4%. Horizontal axis shows 48 SNP markers plant. Left vertical axis shows individual plants used in this study, Right axis shows the recovery rate (%) of each individual. Yellow, recurrent parent type homozygote; Green, heterozygote; White, unknown.
BC2F1 세대의 유전자형 분석
BC1F1 세대에서 선발한 N4 식물체는 RPG 회복률이 87.2%로 표현형 조사에서 MR10-3211 계통과 매우 유사하여 BC2F1 종자를 육성하였다. 총 92개 중 51개 식물체는 AAD 마커의 유전자형이 Aa으로 확인되었다(Fig. 5, Table 2). Background 선발을 위해 BC2F1 세대에서 foreground 선발을 통해 얻은 51개 중에서 48개의 BC2F1 식물체를 대상으로 RPG 회복 정도를 조사한 결과는 87.8%에서 97.8% 범위를 보였다(Fig. 6). 또한 AAD 마커에 의해 선발한 T2-517 공여친 유래 단편은 염색체 2번에 속해 있었으며, 1번, 3번, 5번, 7번, 10번 염색체의 segment를 제외하고 선발된 식물체들에서 거의 대부분이 회복되었다(Fig. 4). 선발한 48개의 BC2F1 식물체들을 대상으로 농업형질을 조사하였다. 그 중 M48 식물체의 과중, 과고, 과폭, 경도 및 당도에서 각각 235.2, 54.7, 64.9, 0.95, 5.2로 반복친 MR10-3211 계통과 가장 유사하였다(Table 4). RPG 회복률이 가장 높으며 농업형질이 반복친 MR10-3211 계통과 가장 유사한 M48 식물체를 이용하여 자가수정을 통해 BC2F2 종자를 육성하였다(Table 4, Suppl. Fig. 2s).
Table 4. Agro-morphological trait performance of BC2F1 individuals derived from MR10-3321 (recurrent) × T2-517 (donor)
BC2F2 및 BC2F3세대에서 유전자형 분석
BC2F1 세대에서 선발한 M48 식물체유래 BC2F2 세대 종자들은 AAD 마커로 유전자형을 분석한 결과, AA:Aa:aa가 1:2:1로 분리되었다. 각각의 유전자형을 가진 식물체를 선발하여 성숙할 때까지 생장시킨 후, 웅성불임과 웅성가임 여부를 조사한 결과 AA 및 Aa형은 웅성가임, aa형은 웅성불임을 보였다. AAD 분자마커에 의해 Aa형, 표현형적으로 웅성가임을 보이는 식물체를 이용하여 BC2F3 세대를 육성하여 웅성불임 엘리트 계통으로 F1 생산에 사용하였다. 따라서 본 연구를 통해 MABC법은 육종연한을 획기적으로 단축시키는 육종기술임이 판명되었다.
고 찰
토마토는 세계에서 가장 중요한 과채류로서 대부분이 F1 품종으로 인공수분에 의해 채종하기 때문에 종자생산 가격이 높고, 자가수정된 종자 혼입에 따른 F1 순도에 문제가 되고 있다(Bhatt et al., 2001; Atanassova, 2007; Su et al., 2012). 따라서 F1 종자 생산을 위해 웅성불임 돌연변이체를 이용하고 있는 실정이다(Atanassova, 2007; Gardner and Panthee, 2010; Panthee and Gardner, 2013). 그 중 ms1035 유전자는 bHLH 전사인자 관련 유전자 프로모터 부분에 398bp 단편이 삽입되어 유전자 발현이 억제됨에 따라 웅성불임성이 나타난 것으로 알려졌으며, 이를 이용하여 제웅 없이 F1 종자 생산체계가 확립되었다(Gorguet et al., 2009, Jeong et al., 2014). F1 종자 생산을 위해서는 우수한 교배모본에 웅성불임성을 도입하여야 하는데, 교배를 통해 ms1035 유전자만 도입된 개체를 선발하는데 많은 시간이 소요된다. 최근 여교배 육종에서 MABC 법이 개발되어 활용되고 있다. MABC 법에서 가장 중요한 것은 양친의 유전자형을 구분할 수 있는 분자표지의 유효성 분석이 매우 중요하다. 본 연구에서 웅성불임성을 선발하기 위해 사용한 연관 마커는 Zhang et al.(2016)에 의해 보고한 녹색 배축을 가진 안토시아닌 결손 관련 AAD 분자표지를 이용하였다. AAD 마커는 웅성불임 유전자와 매우 밀접하게 연관되어 있어, 교배 후대에서도 분리되지 않고 함께 유전되며, F2 세대에서 멘델 유전양식에 입각하여 단일 열성유전자로 잘 분리되었다(Fig. 1). 또한 AAD 마커와 웅성불임성이 밀접하게 연관되어 있는지를 알아보기 위해 F2 세대에서 Aa 유전자형을 가진 식물체를 자가수정하여 후대종자에서 웅성불임개체의 출현을 확인하였다(데이터 미제시). 따라서 AAD 분자마커 기반으로 BC1F1, BC2F1, BC2F2, BC2F3 세대에서 foreground 선발을 수행하였다(Figs. 2 and 5). 지금까지 여교배 육종에서 background 선발을 위한 SNP 마커 세트 선정을 위해 많은 노력을 수행하였다(Herzog and Frisch, 2011; Venkatesh et al., 2018). Background 마커는 어떤 특정 여교배 세대에 있어서 RPG가 어느 정도 회복되는지 여부를 잘 알려주며, 각 세대에서 예상할 수 있는 최상의 여교배 자손에 대해 선발하는 것이 가능하다고 하였다(Herzog and Frisch, 2011; Kim et al., 2013). 또한 표적 유전자좌 이외의 반복친 게놈이 회복된 개체를 선발할 수 있기 때문에 여교배 세대를 대폭 줄일 수 있는 장점을 가진다(Herzog and Frisch, 2011; Kim et al., 2013; Lee et al., 2018). 본 실험에서 background 선발을 위한 SNP 마커세트는 resequencing data를 토대로 양친간 SNP를 토마토 게놈 전체에 균일하게 분포하게 하여 총 48개를 선발하여 사용하였다(Table 3, Suppl. Fig. 1s). 이들 결과는 Tanksley et al.(1996)이 보고한 컴퓨터 시뮬레이션의 이론적인 연구와 거의 일치하는 경향을 보였으며, background 선발 효과는 여교배 초기세대에서 가장 효율이 높다고 하였다(Collard and Mackill, 2008). Background 선발은 염색체상의 거의 동등한 거리로 약 20cM 정도면 좋은 결과를 얻을 수 있다고 하였다(Randhawa et al., 2009). 따라서 본 실험에서는 background 마커의 거리가 약 15-23cM으로 설정하였다. MABC 프로그램의 효율을 결정하는 또 다른 중요한 요인은 도입하려고 하는 표적 유전자수, 유전자 지도, 교차율 및 적용되는 선발 전략이라고 하였다(Servin and Hospital, 2002; Collard and Mackill, 2008). 본 연구에서는 이런 측면에서 볼 때 매우 단순한 열성유전자를 표적으로 하였으며, 선발은 매우 밀접하게 연관되어 있는 마커를 이용하였기 때문에 선발 전략이 좋았다고 생각된다. 본 실험에서는 BC1F1 세대의 48개체 중에서 N3, N4 및 N20 개체가 RPG 회복률이 87.2%로 가장 높았다. 그러나 Randhawa et al.(2009)은 BC1F1 200 개체를 background 선발하였을 때 RPG의 회복률이 97.5%인 개체를 얻었다. BC2F1 세대의 각각의 식물체는 과중, 과장, 과폭, 경도 및 당도 등의 농업 특성검정을 통해 최종 선발하였다(Table 4, Suppl. Fig. 2s). 또한 BC2F1 세대의 모든 개체에서 T2-517 donor친 유래 단편은 염색체 1번, 2번, 3번, 5번, 7번 및 10번에서 이형접합체로 존재하였다(Fig. 4). 특히, 10번 염색체에서 이형접합형이 높은 이유는 T2-517 공여친 유래 농업형질관련 단편이 계속 남아있기 때문이라고 생각된다.
본 실험에서는 MABC에 의해 웅성불임 엘리트 계통 M48 식물체가 최종 선발되었으며, F1 종자생산을 위해 BC2F2 및 BC2F3 세대를 진전시켜 교배모본으로 활용하였다. 현재의 데이터로 볼 때 MABC 육종 프로그램은 세대단축을 위한 획기적인 육종법으로 판단된다.
Supplementary Material
Supplementary materials are available at Horticultural Science and Technology website (https://www.hst-j.org).
- HORT_20190074_Table_1s.pdf
Primer information for using a high-throughput genotyping system (EP1TM, Fluidigm)
- HORT_20190074_Fig_1s.pdf
Position of the 48 MABC markers on tomato chromosomes. Bars represent the twelve tomato chromosomes, while the length of each chromosome (Mb) is indicated below each bar. Chromosomal regions are shown on the left side. On the right, the names and locations of the 48 MABC markers used are displayed in red rings.
- HORT_20190074_Fig_2s.pdf
Agro-morphological fruit traits in BC2F1 population for male-sterility MR10-3211 line of the tomato.